Bioinformatik
Contents
Status & Planer
Generelt er forløbet og dennes beskrivelse lavet for at gøre prøve; dels med hensyn til at udforske, hvordan man overhovedet bedriver "CT i fag" med et konkret eksempel; dels mht. brug af meget tidlig udgave af en skabelon for forløbsbeskrivelser.
Evaluering af forløbet:
- Forløbet er afprøvet i en 1. G klasse i Bioteknologi på Silkeborg Gymnasium i Maj 2016. Med ret god succes, synes vi selv :-)
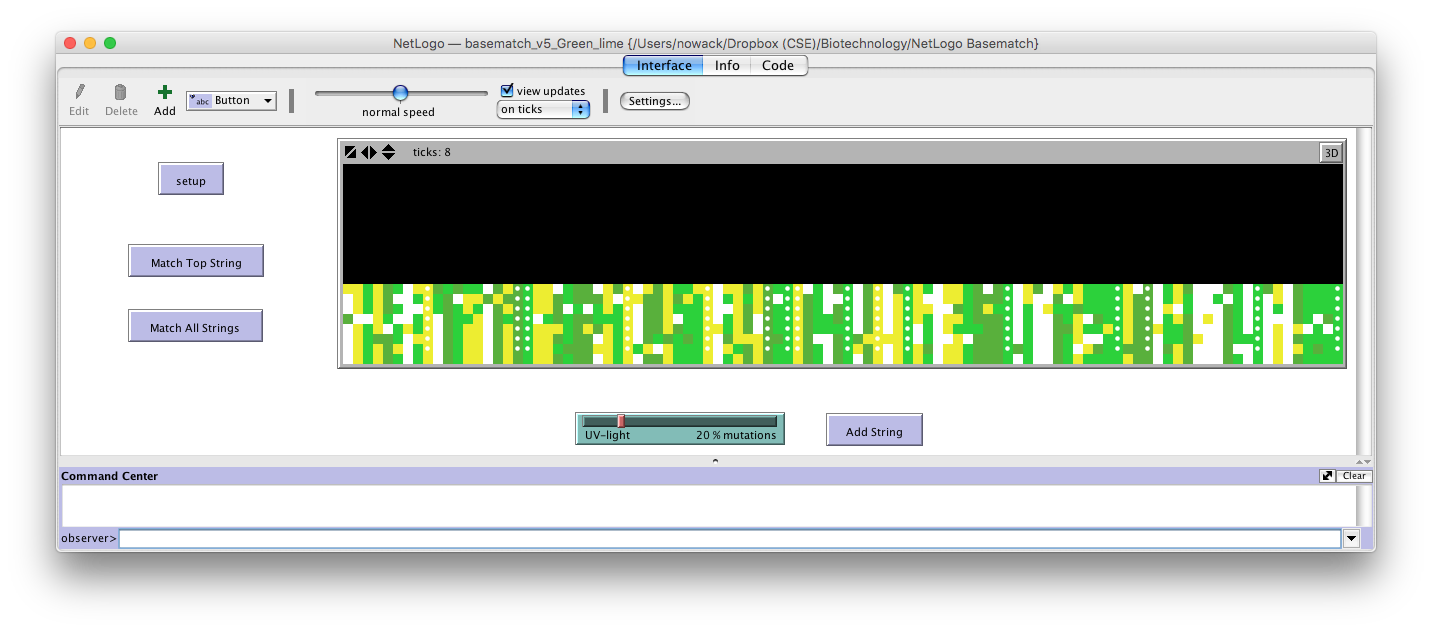
- NetLogo modellen (selve simuleringen) lider under det faktum, at den ikke er specielt agent-orienteret. Agenterne er således passive "felter" og har dels ikke egl. autonom adfærd; dels er simuleringen ikke tidsafhængig (da det ikke giver mening i denne kontekst).
- Når man anvender NetLogo får man grafisk brugergrænseflade og interaktionsdesign foræret.
- Forløbet er beskrevet yderligere (men meget kortfattet!) i en artikel, der pt. er under review.
Evaluering af beskrivelsen:
- Dette eksempel på en forløbsbeskrivelse er lavet med udgangspunkt i denne skabelon.
- Det er forvirrende med både inline figurer og henvisning til powerpoints.
- Det er forvirrende at samlingen af materialer ikke kan ses og overskues samlet, men er scattered all over beskrivelserne af aktiviteterne. Men man har nok brug for begge "views" på materialer: dels i aktivitets-kontekst; dels for sig selv, samlet.
- Der bør nok tilføjes mere tekst og forklaring til beskrivelsen af aktiviteterne.
- Videoerne under Reflektionsafsnittet indeholder to korte optagelser af eleverne fra prøveforløbet, hvorfor dette er passwordbeskyttet, og ikke må videredistribueres/anvendes uden forangående kontakt til Line. Password kan fås ved henvendelse til Palle.
- Undervejs bliver man usikker på formålet af disse forløbsbeskrivelser. Er det...
- interne arbejdsdokumenter til arbejdsgruppen af materialeudviklere?
- beskrivelser af forløb, der skal kunne tages og anvendes direkte af andre undervisere?
- beskrivelser der skal kunne anvendes af elever - f.eks. i et flipped-classroom set-up?
Det er ikke planen at gøre frygteligt meget mere ved denne beskrivelse. Alligevel overvejer vi at få det afprøvet af en håndfuld andre lærere på hold og skoler indenfor bioinformatik. Men vores primære fokus fremover er at få lavet et nyt forløb til bioteknologi og/eller biologi, hvor der i højere grad benyttes "rigtige" agenter.
Introduktion
Formålet med forløbet er trefold:
- Generelt at introducere de studerende til udvalgte elementer af computational thinking: abstraktion, modellering og algoritmer (sekvens, iteration og selektion).
- Generelt at arbejde med et centralt emne i bioteknologi vha. en computationel thinking (CT) indgangsvinkel. Her er bioinformatik oplagt. I bioinformatik arbejdes der med at sammenligne DNA-sekvenser fra mange forskellige organismer, hvilket betyder at der ofte arbejdes med store mængder data. Det er derfor praktisk at anvende computerprogrammer til at behandle data og hjælpe de studerende med en relevant tolkning af informationen.
- Konkret - som eksemplifikation af kombinationen af de to ovenstående formål - at lade de studerende arbejde med en simpel repræsentation af DNA sekvenser, der kan genereres automatisk og sammenlignes.
Forløbet strækker sig ialt over 4x75 minutter.
Forudsætninger
- De studerende arbejder med deres egne computere, hvorfor det forudsættes, at de kan installere NetLogo software på denne (alternativt findes der en webbaseret udgave af NetLogo).
- Adgang til små whiteboards, der bruges parvis til at lave modeller.
- Adgang til digital kamera / mobil mht. at uploade fotos af whiteboards.
- Der er ingen faglige forudsætninger indenfor hverken bioteknologi eller CT.
- I løbet af forløbet bliver de studerende bedt om at løse specifikke opgaver i NetLogo og om at gemme deres modeller som filer.
Aktiviteter & Materialer
Forløbet er struktureret i tre logiske klumper: introduktion, arbejdsopgaver og perspektivering.
I det følgende refereres der til disse slides i DropBox: [PPT]
Introduktion:
En kort introduktion til emnet bioinformatik og DNA-sekventering ved brug af TEDEd's nedenstående to videoer og Biotech Academy, DTU (under videoerne).
Introduktion til bioinformatik fra Biotech Academy, DTU.
Link til spørgeskema med arbejdsspørgsmål og evalueringsspørgsmål.
- Arbejdsspørgsmål til teksten fra Biotech Academy, DTU, om bioinformatik (slide 1 og 2).
Perspektivering:
- Problemformulering: Til slut bliver de studerende bedt om at udarbejde problemformuleringer, hvor deres opnåede viden om NetLogo-simuleringen og bioinformatik skal integreres med dansk- eller historiefaget. Disse formuleringer bruges til evaluering. Se eksempel i Reflektionsafsnittet.
- De studerende er på forhånd blevet introduceret til ”Berkeley modellen”.
Refleksioner over designet af forløbet
Under aktiviteten "Arbejdsopgaver" anvendes det didaktiske princip "Use-Modify-Create", idet eleverne starter med at lave en model (rutediagrammet) af en udvalgt stump kode. Senere beder vi dem løse konkrete opgaver, ved at ændre specifikke dele af koden.
Der var gode erfaringer med at lade de studerende "lege" med den specifikke simulering i NetLogo, forholdsvis tidligt i forløbet. I videoklippet nedenfor ses en studerende begejstring over at have lagt flere DNA-sekvenser over hinanden med samme grad af mutationer i hver af dem.
I forbindelse med de stillede arbejdsopgaver til ændringer i kode-delen af simuleringen, har to studerende foretaget ændringer i koden som ikke er en del af løsningen af opgaven. De identificerer selv ændringen og forklarer at den ikke er relevant i bioteknologisk sammenhæng, men at den kunne være brugbar i en anden sammenhæng. De bevæger sig altså mellem overvejelser omkring det bioteknologisk faglige indhold i simuleringen og overvejelser over hvad der er muligt i koden. Se videoen nedenfor.
Introduktion til symboler og rutediagrammer blev givet forsøgsvist for at de studerende kunne anvende et "sprog" til at beskrive aktiviteter i koden bag simuleringen. Det var forholdsvist nemt for de studerende at anvende symbolerne til at beskrive et kendt eksperiment, men betydeligt mere udfordrende at at anvende symbolerne i forbindelse med et stykke ny kode.
Målopfyldelse er forsøgt dokumenteret ved bl.a. at analysere de studerendes brug af symboler og rutediagrammer. Også problemformuleringerne med integrering af simuleringen i emnet bioinformatik kan give et indblik i dette. Se eksempel nedenfor.
Endelig tilgodeser simuleringen af DNA-sekvenser, og sammenligningen af disse, fagets læreplan som påpeger at de studerende skal introduceres for fagets forsknings- og anvendelsesaspekter.